论文总字数:16928字
目 录
- 前言……………………………………………………………………1
- 材料与方法
2.1地点介绍…………………………………………………………………………………2
2.2 FACE系统………………………………………………………………………………2
2.3土壤采样和土壤特性分析………………………………………………………………2
2.3.1土壤DNA提取……………………………………………………………………3
2.3.2实时定量PCR……………………………………………………………………3
2.3.3高通量测序步骤…………………………………………………………………3
2.4高通量测序数据的处理…………………………………………………………………3
2.5共生网络的构建和可视化………………………………………………………………4
2.6数据分析…………………………………………………………………………………4
- 结果
3.1土壤中的可溶性有机碳(DOC)和有效氮(AN)………………………………………4
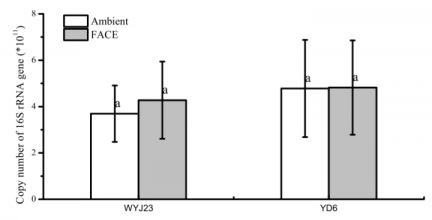
3.2细菌16S rRNA基因的基因拷贝数………………………………………………………5
3.3细菌群落的α-多样性指数………………………………………………………………5
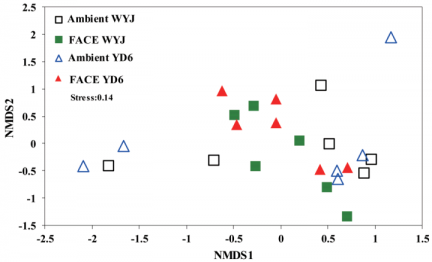
3.4细菌群落结构 ……………………………………………………………………………6
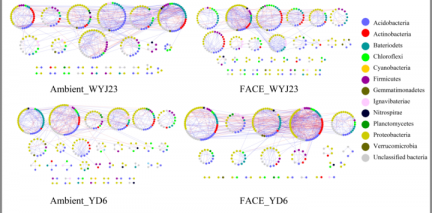
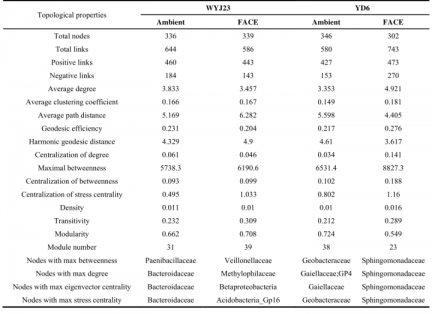
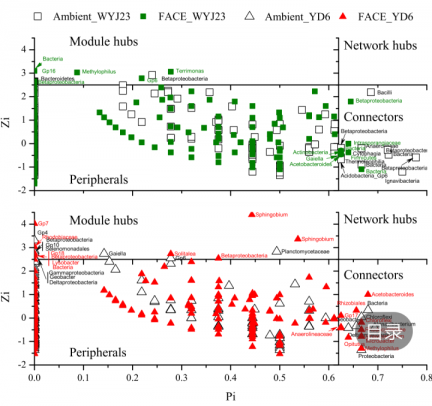
3.5共生网络模式 ……………………………………………………………………………6
3.6细菌种系共现的不同反应 ………………………………………………………………7
- 讨论
4.1水稻品种引起土壤不稳定组分的变异…………………………………………………8
4.2土壤细菌群落多样性没有明显变化……………………………………………………9
4.3 eCO2下水稻品种可以改变土壤细菌类群之间的相互关系……………………………9
- 结论……………………………………………………………………10
- 参考文献………………………………………………………………11
FACE条件下不同水稻品种对土壤微生物群落的影响
成瑾
摘要:CO2浓度升高(e CO2)对农业生态系统的影响一直备受关注。但,目前对土壤微生物群落特性在eCO2条件下对不同植物品种的响应尚不明确。本研究中,我们选取两种水稻品种--CO2耐受品种武运粳23(WYJ23)和CO2敏感品种扬稻6(YD6)--研究其在eCO2条件下对土壤细菌群落的影响,采用实时荧光定量PCR(qPCR)和高通量测序表征细菌群落。用共生网络分析揭示细菌物种之间的生态相互作用关系。 结果显示,在不同处理下,尽管eCO2和水稻品种对细菌群落的α多样性指数及细菌群落结构方面没有显著影响,但显著改变了微生物群体中的共生网络模式。相比WYJ23,在eCO2背景下的YD6有更多的群落联系,但模块化程度更低、集中度更高, 这可能会导致生态网络的不稳定。此外,更多的变形菌和酸杆菌在YD6处理中成为关键菌种,而在WYJ25处理中变化不明显。总体而言,我们的研究揭示了在短期eCO2的背景下,高应答水稻品种可以影响细菌群落交互性而不是土壤微生物多样性和群落结构,这在CO2-植物-土壤生态系统的关系系统中还有待进一步调查。
关键词:FACE,水稻品种,共生网络分析,16SrRNA基因
Abstract:The influence of elevated CO2 concentration (eCO2) on agricultural ecosystems has been addressed in recent years. However, there is still little concern on soil microbiome properties in response to different plant cultivars under eCO2 condition. In this study, two rice cultivars, i.e. the CO2-tolerant cultivar Wuyunjing23 (WYJ23) and the CO2-sensitive one Yandao6 (YD6), were selected to investigate the soil bacterial response to eCO2. Real-time qualitative PCR (qPCR) and high throughput sequencing were used to characterize the bacterial community. Co-occurrence network analysis was employed to reveal the ecological interactions between bacterial species. Results showed, although no significant differences among all treatments in terms of bacterial population, alpha-diversity indices, and the bacterial community structure, eCO2 and rice cultivars markedly changed the co-occurrence network patterns in microbiome. Specifically, compared to WYJ23, YD6 under eCO2 had more links, but lower modularity and greater centralization degree, which might induce instability of the ecological network. Besides, more Proteobacteria and Acidobacteria severed as keystone bacteria in YD6 compared to those in WYJ25. Collectively, our study unraveled the CO2-responsive rice cultivars could influence the bacterial interactions rather than soil bacterial diversity and structure under short-term eCO2, which is indicative for further investigation on the relationship of CO2-plant-soil ecosystems.
Keywords: free air carbon dioxide enrichment, rice cultivar, co-occurrence network analysis, 16SrRNA gene
1.前言
大气中的二氧化碳浓度([CO2])在接下来几十年预计会持续上升[1],到本世纪中叶,二氧化碳浓度从现在的370ppm上升到550ppm[2]。越来越多的证据表明全球生态系统,如草地,森林和农业生态系统会因二氧化碳(eCO2)升高而发生改变[3]。陆地生态系统的结构和功能在很大程度上受到土壤微生物的影响,要全面评价大气二氧化碳上升对植物以及整个陆地生态系统的影响,必须首先弄清土壤微生物的变化。由于二氧化碳升高促进了土壤中有机碳的输入,进而为土壤微生物提供了更多可降解底物,因而我们猜测促进了微生物的活性。众所周知,eCO2通过刺激植物(如水稻)生长增加植物初级生产量[4]。水稻是世界上最重要的粮食作物,覆盖了地球可耕地的9%。已有很多研究表明在二氧化碳浓度上升条件下水稻生态系统各种水稻品种产量变化[5]。然而,由于光合累积作用,水稻在eCO2条件下产量增幅(9-15%)远低于预期(30%)[6]。因此,科研工作者着力于在各水稻品种中选育出对二氧化碳具有敏感性即CO2高应答的品种水稻。在中国开放式空气二氧化碳浓度增高平台(FACE)下,已有研究人员发现了一种籼稻基因型扬稻6号(YD6),在eCO2条件下能够达到接近预期的籽粒产量(比正常大气二氧化碳浓度高出约30%),因而该品种具有较高的应用前景。
尽管在eCO2对作物影响方面已经达成共识,然而在土壤微生物群落方面仍存在争议。对于微生物功能或群落结构组成存在或正[7]、或负[8]、或者无影响[9]。例如Wang等人发现在秸秆还田条件下,FACE对于产甲烷菌在水稻分蘖期有促进作用,而在抽穗期和收获期有抑制作用,并且在低N条件下可以达到显著性水平,秸秆不还田时无明显刺激作用。另外,已有研究人员发现,二氧化碳浓度升高对细菌数量的影响较为明显,而对真菌数量影响不大,这可能与植物根迹分泌物有关。土壤微生物被广泛认为对陆地生态系统的功能至关重要。它们在土壤中参与多种生物地球化学作用,对环境中的扰动很敏感[10]。从这个意义上说,土壤各种微生物对eCO2的不同反应可能是由于土壤类型,植物群落等的差异造成的。考虑到上述提到的高产量水稻品种YD6,我们相信研究在eCO2条件下生长的不同水稻品种的微生物反应,这些信息将会对于建立高产量的水稻生态系统有所帮助。
值得注意的是,迄今为止关于eCO2效应的大多数研究集中在群落总体特征(即α-多样性)和种群之间系统发育分类的相对丰度(即β-多样性)[11]。该类分析是有助于解释微生物群落在土壤中的结构,但鉴于土壤生态过程的复杂性,对微生物种群之间的相互作用过程(直接或者间接)缺乏研究[12]。共生网络分析被广泛用于研究全球变化对植物和动物多样性影响的方法,可加深为微生物之间的关联模式的理解[13]。基于共生网络分析,我们可以获得关于不同微生物类群之间的积极的(共生和互助)或消极的(捕食和竞争)相互作用以及对于网络稳定化有重要作用的关键种的进一步了解,也有助于确定某些微生物的功能作用。随着高通量测序技术的发展,海量的微生物序列数据为研究共生网络提供可能,这些研究可能超越常规的α-和β-多样性分析。事实上,这样的分析已在早期研究中被应用于评估微生物对eCO2的反应[14]。从文献中可以看出,网络分析得到的拓扑参数可以反映网络的稳定性,如模块化或分散程度[15]。然而,在eCO2条件下植物品种是否影响细菌交互网络的稳定性至今依然不清楚。
在本研究中,我们推测水稻品种在eCO2条件下会使土壤细菌类群共生模式产生较大的改变,对其共生模式的影响大于对群落多样性和结构的影响。为此,我们选取了两种水稻品种:CO2敏感品种YD6和耐CO2品种武运粳23(WYJ23),研究其在FACE条件下对土壤微生物群落结构及共生网络的影响。该实验始于2014年,在连续种植的第三年即2016年,选择在水稻开花期采集FACE平台中和大气环境中对照组水稻的土壤样品。用实时荧光定量PCR和高通量测序技术检验土壤细菌群落的变化,用共生网络分析揭示不同细菌类群之间的交互关系。
2.材料与方法
2.1.地点介绍
FACE系统成立于中国江苏省江都市(119°42'0"E,32°35'5"E)。土壤被归类为冻土花粉。相关的土壤性质如下:9.2%砂土(1-0.05mm),65.7%淤泥(0.05-0.001mm),25.1%粘土(lt;0.001mm),1.2g/cm3堆积密度,15.0g/kg土壤有机C(SOC),1.59g/kg总氮,1.23g/kg总磷,10.4mg/kg可用磷,pH6.8。该站点位于亚热带气候带,年平均降水量900-1000毫米,年平均气温16摄氏度,平均日积分辐射量12.3兆焦耳/平方米,年日照总时间超过2000小时,无霜期超过230天。
2.2.FACE系统和耕耘模式
FACE系统中,有三个二氧化碳浓度(200±40μmol/mol-1)高于环境温度(355±15μmol/mol-1)的八角环(以下简称eCO2环)和3个无CO2增加的大气环境环(以下简称aCO2环)。所有直径12.5米的eCO2环都远离aCO2环90米以上。高压下的纯二氧化碳从农作物周围的管子朝向eCO2环的中心全天被释放到作物冠层上方约50厘米处。
为了研究不同品种对eCO2的反应,本研究中培育了两个水稻品种:耐CO2近交籼稻品种“武运粳23”(WYJ23)和CO2敏感杂交栽培品种“扬稻6号”(YD6)。在以往的研究中,YD6已被证明对eCO2敏感,与大气环境环境CO2处理(aCO2)相比,eCO2条件YD6的下籽粒产量增加32.9%;相比之下,WYJ23的增幅仅为12.8%。关于不同水稻品种的试验开始于2014年。由于两个水稻品种不同的分蘖能力,这些水稻品种的苗被人工移植到种植密度为每丘陵一棵植物的大气环境和每丘陵两棵植物的FACE环境的地块中。丘陵的间距为16.7厘米×25厘米(相当于每平方米24丘陵)。对于所有处理,N用作基肥(占总量的40%)。移栽前1天,在分蘖早期作为追肥(占总量的30%)和穗起始(PI)阶段(占总量的30%),以22.5克N每平方米的密度进行施肥。磷(P)和钾(K)以9克P2O5m-2和9克K2Om-2作为复合肥施用; P和K在移栽前1天都作为基肥施用。
2.3.土壤采样和土壤特性分析
在连续种植的第三年(2016年),我们在开花期收集了eCO2和aCO2环的每个栽培品种子区的两份样品。也就是说,共有24个样本。采样前,田间排水5天以方便采样,并在0〜15厘米深度取3.5厘米直径的核心土样。从每个子区域随机取5个土芯样品,然后混合在一起制成一个复合样品。随之将样品分成两个子部分:一个保存在4℃用于生化测定,另一个保存在-80℃用于分子分析。
溶解的有机碳(DOC)和有效氮(AN)从10克鲜土中萃取出来,使用50ml0.5mol/lK2SO4超纯水进行处理再进行离心(8000rpm,10分钟)。用碳氮分析仪(Elementar,德国)和连续流量分析仪(Skalar,荷兰)分别对通过0.45mm滤膜的滤液进行分析。
2.3.1土壤DNA提取
根据说明书,使用FastDNA SPIN Kit for Soil(MP Biomedicals,Santa Ana,CA)从新鲜土壤(0.5g)中提取土壤DNA。细胞溶解是在速度设定为6.0的FastPrep®24珠击仪器中进行剧烈振荡并持续40秒。将提取的DNA溶解在70μl的DNA洗脱液中并在-20°C保存。使用Nanodrop ND-1000 UV-Vis分光光度计测定DNA的量和纯度。
2.3.2实时定量PCR
在CFX96Tm Real-Time系统(Bio-Rad,USA)上通过实时定量PCR测量(qPCR)细菌16SrRNA基因片段的丰度。量化是使用引物组519F/907R(519F:5'-CAGCMGCCGCGGTAATWC-3',907R:5'-CCGTCAATTCMTTTRAGTTT-3')在20μl1×SYBR®Premix Ex Taq Tm反应混合物(TaKaRa,大连,中国)对细菌16SrRNA基因进行定量测量,一式三份。用10倍连续稀释的质粒DNA制作标准曲线。扩增产物的特性通过熔解曲线分析来确定。
2.3.3高通量测序步骤
用通用引物519F/907R扩增土壤细菌16SrRNA基因,标记V4-V5高变区。用MiSeq测序系统(Illumina Inc.,USA)进行测序,对于每个重复DNA样品,用独特的5-bp条形码对引物进行标记。在含有各个浓度为1.25μM的脱氧核苷三磷酸,2μL(15μM)正向和反向引物,2个单位(即400ml)DNA聚合酶(TaKaRa,日本)的50μL反应混合物中进行PCR反应,并且各反应混合物中有1μL(50ng)基因组群DNA作为模板。热循环如下:95℃45秒,58℃45秒,72℃1分钟,最后72℃延伸7分钟,循环35次。包括使用无菌水代替土壤DNA的阴性对照以检查引物或样品DNA污染。每个DNA样本在三个技术重复中扩增。然后合并反应产物,用QIA高速PCR纯化试剂盒(QIAGEN)进行纯化,并使用NanoDrop ND-1000(Thermo Scientific,USA)进行定量。各样品的条形码PCR产品等摩尔量归一化,然后使用TruSeqTM DNA样品制备试剂盒制备,并使用MiSeq平台上的MiSeq试剂盒(500-cycles-PE)依照制造商协议进行测序。
剩余内容已隐藏,请支付后下载全文,论文总字数:16928字
相关图片展示: